Python实现PCA降维算法
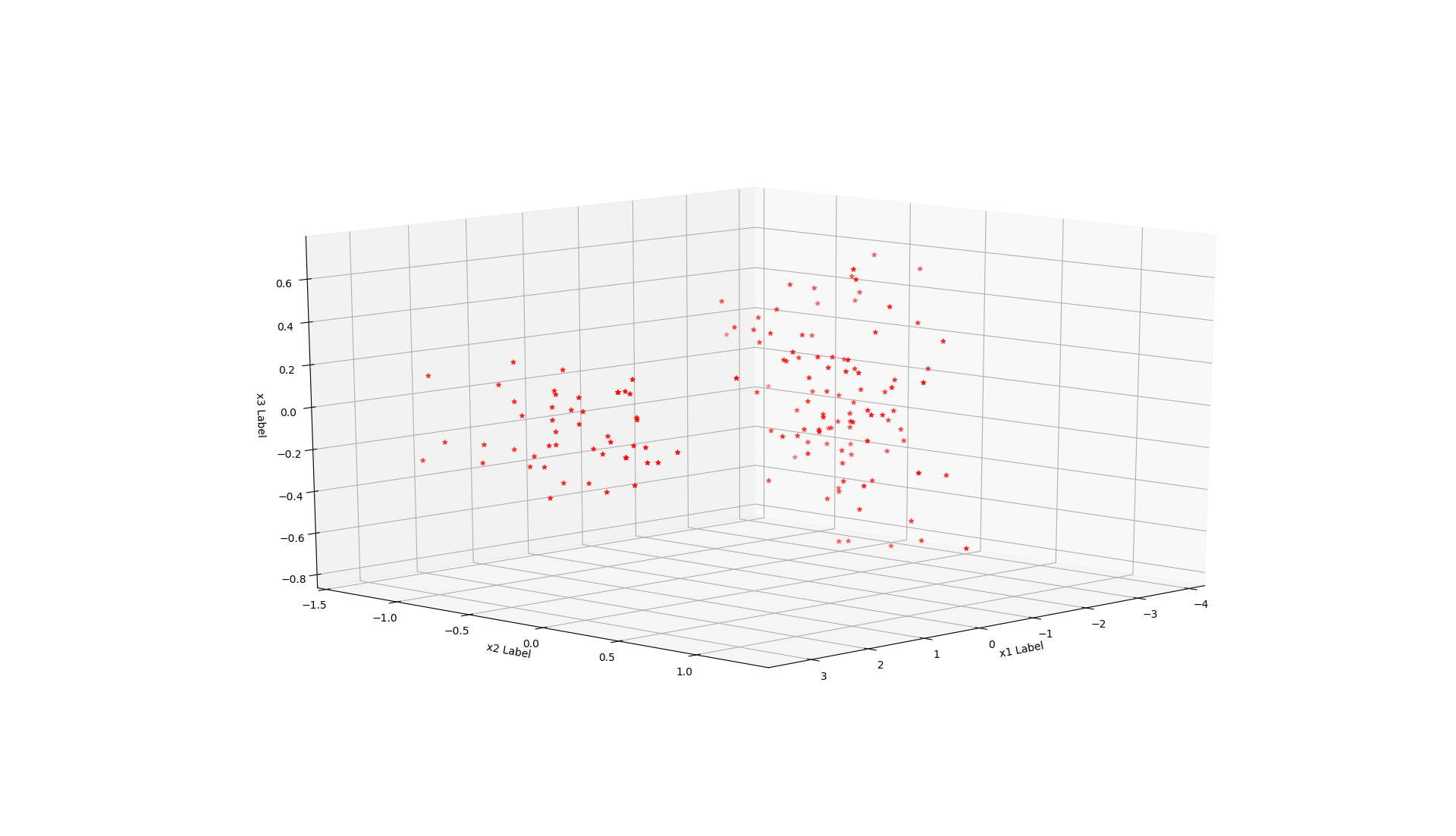
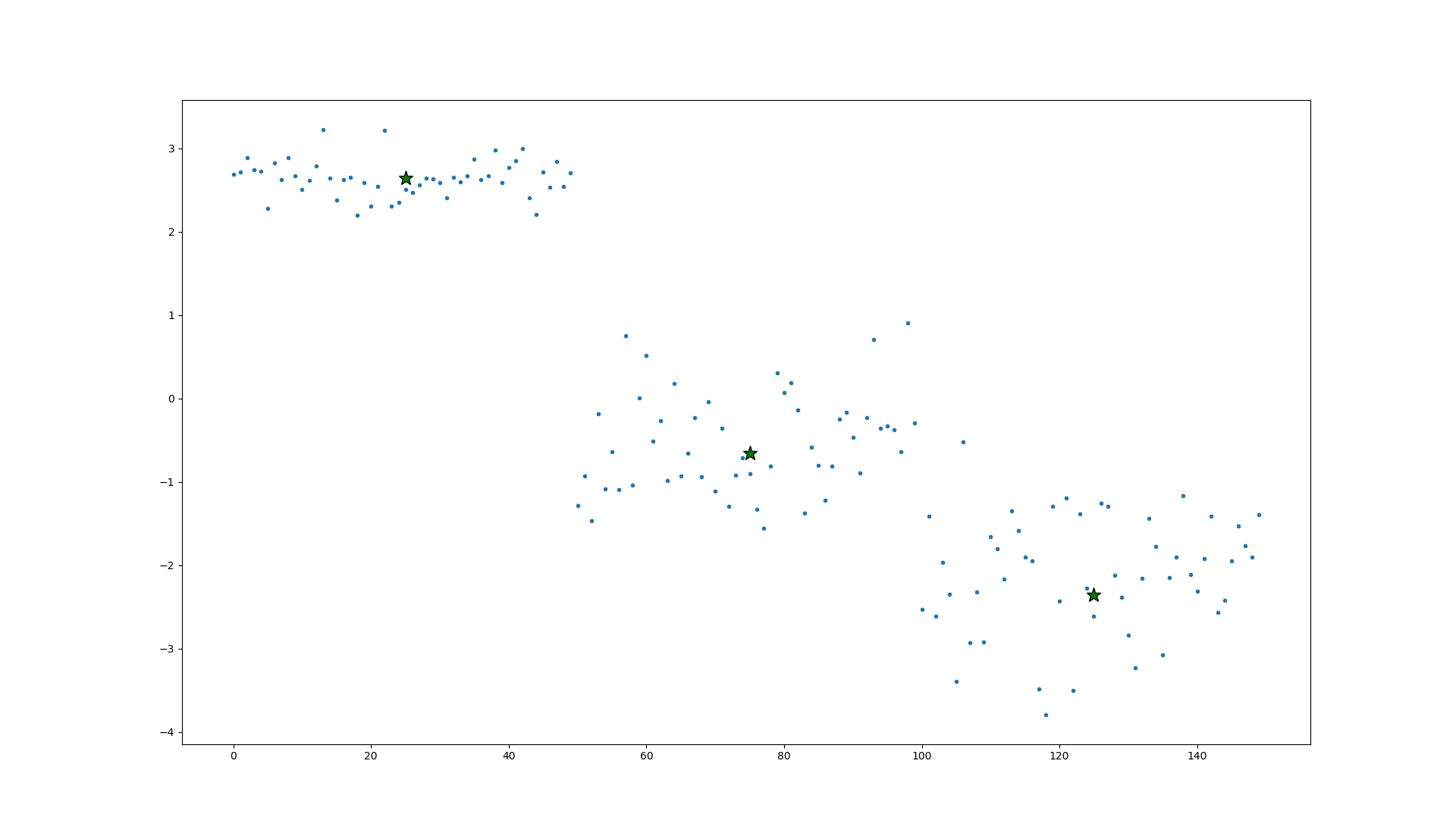
这是第二个无监督学习的算法,是一个降维算法,可以把多个特征进行压缩,我在压缩后计算了与原数据的偏差,当我把四个特征压缩为三个时偏差只有0.5%,压缩为一个特征时偏差也只有7%,当只有一个特征时把数据展开也可以轻易的分为三类,所以这是一个非常优秀的算法。
值得注意的点是在计算奇异矩阵时遇到的问题,首先我们有一个m×n(m个数据,n个特征)的矩阵X,我们希望得到一个m×k的矩阵Z,具体降维过程分三步:
·第一步:均值归一化,就是把每一个数都减去总数的平均值,得到的一个和平均数差距的新矩阵Xj。
·第二部:计算协方差矩阵,在这里要注意的时,Xj(i)是一个n×1的矩阵,Xj(i)的转置是一个1×n的矩阵,所以他俩相乘得到一个n×n的矩阵Σ,其实就是的到一个奇异矩阵,因为只有奇异矩阵才可能有特征值。
·第三部:奇异值分解,计算∑的特征值,使用svd()函数分解出U,S,V三个向量,U也是一个n×n的矩阵,在U中选取k个向量,获得一个n×k的矩阵Ureduce,新的特征矩阵z就等于Ureduce的转置(k×n)乘以X(n×m)结果得到一个k×m的新矩阵
#!/usr/bin/python
# coding=utf-8
from sklearn.datasets import load_iris
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
import numpy as np
from sklearn.cluster import KMeans
class MYPCA:
def __init__(self, data, k):
self.m = len(data) # 训练数据个数
self.n = len(data[0]) # 现在的特征数
self.k = k # 优化后的特征数
self.X = data
# 第一步是均值归一化。我们需要计算出所有特征的均值
def data_preprocess(self):
sum = 0
for i in self.X:
sum += i
u = sum/self.m
self.newX = np.empty([0, self.n])
for i in self.X:
self.newX = np.row_stack((self.newX, i - u))
return self.newX
# 第二步计算协方差矩阵 传入均值归一化后的矩阵 Σ=1𝑚Σ(𝑥(𝑖))𝑛𝑖=1(𝑥(𝑖))𝑇
def covariance_matrix(self, X):
sum = 0
for i in X:
i = i[np.newaxis, :]
sum += np.dot(i.T, i)
sigma = sum/self.m
return sigma
# 计算新的特征向量Z
def get_z(self, U, X):
z = np.empty([self.k, 0])
Ureduce = U[...,0:self.k]
for i in X:
i = i[np.newaxis, :]
t = np.dot(Ureduce.T, i.T)
z = np.column_stack((z, t))
return z
# 计算训练集误差
def error_analysis(self):
S = self.S
sigmaK = 0
sigmaN = 0
for i in range(self.n):
if i < self.k:
sigmaK += S[i]
if i < self.n:
sigmaN += S[i]
return 1 - sigmaK/sigmaN
# 恢复到之前维度
def rovecor_dimensional(self):
Ureduce = self.U[..., 0:self.k]
Xappox = np.dot(Ureduce, self.z)
return Xappox
def train(self):
newX = self.data_preprocess()
sigma = self.covariance_matrix(newX)
self.U, self.S, self.V = np.linalg.svd(sigma)
# 这里使用均值归一化后的X和原X对结果没有影响
#self.z = self.get_z(self.U, self.X)
self.z = self.get_z(self.U, newX)
return self.z.T
# 构造训练集:引入鸢尾花数据集来作为训练集, 具有四个特征,分三类
iris = load_iris()
data = iris.data
data = np.array(data[:])
m = len(data)
#np.random.shuffle(data)
# 把四个特征压缩为三个
irispca = MYPCA(data, 3)
z = irispca.train()
error = irispca.error_analysis()
print(error)
x1 = z[:, [0]]
x2 = z[:, [1]]
x3 = z[:, [2]]
fig = plt.figure()
ax = fig.add_subplot(111, projection='3d')
ax.scatter(x1, x2, x3, c='r', marker='*')
ax.set_xlabel('x1 Label')
ax.set_ylabel('x2 Label')
ax.set_zlabel('x3 Label')
plt.show()
# 把四个特征压缩为一个
irispca = MYPCA(data, 1)
z = irispca.train()
plt.plot(z, '.')
error = irispca.error_analysis()
print(error)
# 使用Kmeans的算法验证一下是否还可以正确分类
kmeans = KMeans(n_clusters=3, random_state=0).fit(z)
kmeans_u = kmeans.cluster_centers_
u = np.transpose(kmeans_u)
plt.plot([m/6, m/2, 5*m/6], u[0], '*', markerfacecolor='g', markeredgecolor="k", markersize=14)
plt.show()